About Us
随着以深度测序为代表的高通量生物技术在生命科学领域的广泛应用,各种生物学大数据以指数增长大量涌现。这些数据之中蕴藏着大量的宝藏,即生物学的新规律、新发现。但是,这些海量的、指数增长的、并且高噪声的生物数据也带来了巨大的数据分析技术上的挑战。
课题组研究围绕细胞调控图谱解析这一核心科学问题,以生物信息学分析技术、方法与平台开发为基础,通过综合运用大数据与统计学习(statistical learning)等计算方法,整合高通量遗传学与功能基因组学数据,致力于在单细胞水平精准解读基因组功能。在国家科技部、国家自然科学基金委、国家教育部等多年的持续支持下,课题组目前拥有总计算能力逾千亿次的高性能计算平台、高可靠海量存储设备、完备的分子与细胞实验平台等,为科研工作提供全面完善的支撑。
我们相信多元化的团队能够激发创意、促进创新。课题组成员来自从数学、计算机到生物、医学等多种背景与专业,组内氛围融洽,同学之间互相帮助。课题组注重人才培养,通过系统、全面的专业训练,为来自不同背景的同学构筑坚实的专业基础。
依托北京大学暨昌平实验室的大力支持,课题组长期招聘研究生、博士后、研究人员等多方面人才,欢迎加入!
News
高歌课题组提出人类转录调控元件建模与相关非编码变异功能解析方法
细胞是生物体生命活动的基本单元,生命活动的正常进行依赖于精确的基因表达调控过程,而基因转录调控过程则是基因表达 Read more about 高歌课题组提出人类转录调控元件建模与相关非编码变异功能解析方法[…]
高歌课题组提出跨平台、多模态空间组学比对与整合方法
生命是细胞有序排列构成的整体。单个细胞在脱离生命体后难以独立发挥功能。因此需要联合细胞所处的微环 Read more about 高歌课题组提出跨平台、多模态空间组学比对与整合方法[…]
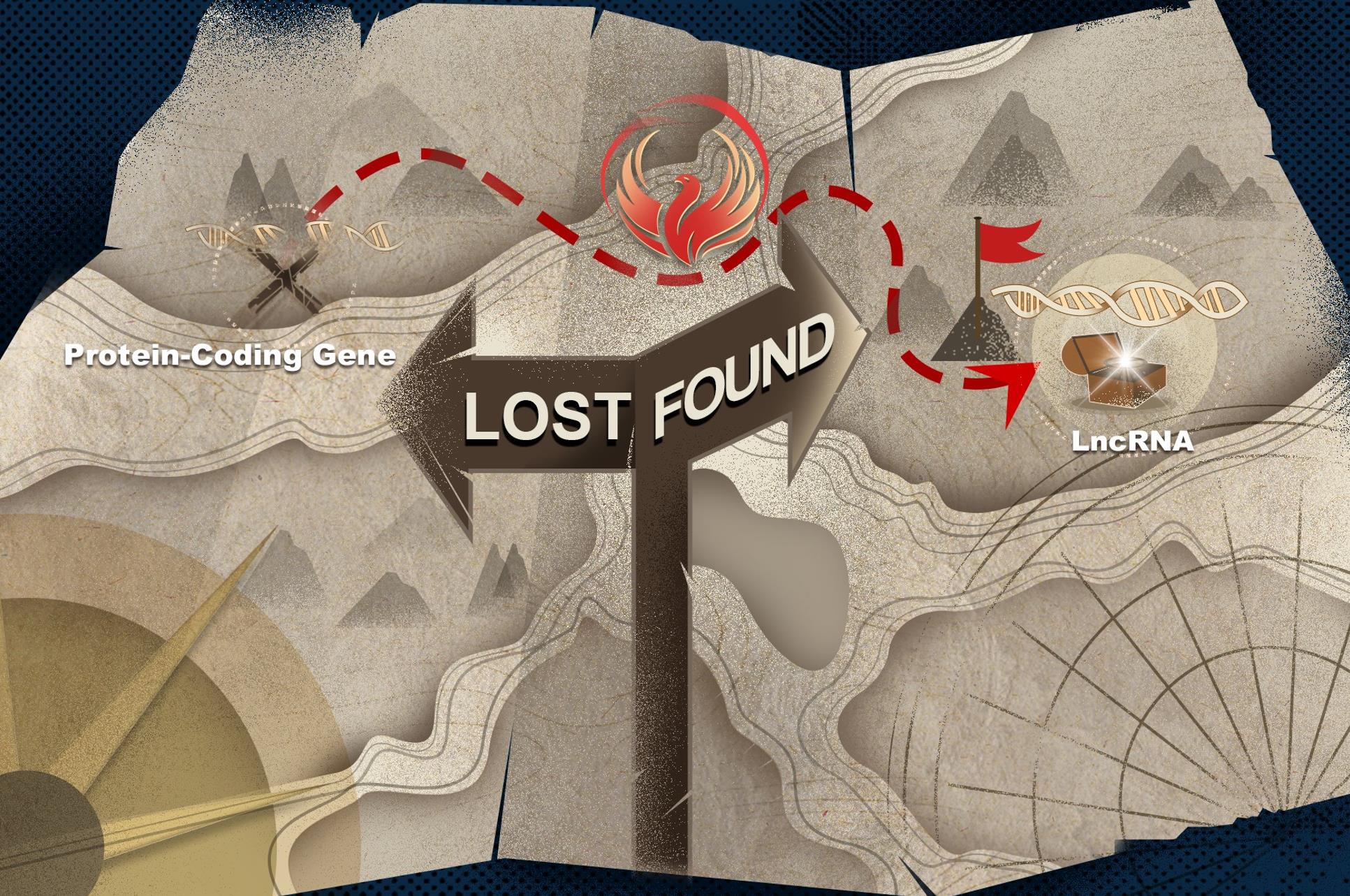
Mol. Biol. Evol. | 高歌课题组提出基因丢失鉴定新方法
通过转录-翻译过程,蛋白编码基因(Protein-coding genes,以下简称基因)可以指导合成对于生命 Read more about Mol. Biol. Evol. | 高歌课题组提出基因丢失鉴定新方法[…]
祝贺GLUE入选2022年度“中国生物信息学十大进展”
高歌研究组2022年发表于《自然·生物技术》(Nature Biotechnology)杂志的研究成果《基于图 Read more about 祝贺GLUE入选2022年度“中国生物信息学十大进展”[…]
Nat. Biotechnol. | 高歌课题组提出单细胞多组学数据整合与调控推断新方法
基因的转录在生物学中心法则中处于承上启下的重要环节,与相对“静态”的基因组相比,转录组在不同组织/器官/发育阶 Read more about Nat. Biotechnol. | 高歌课题组提出单细胞多组学数据整合与调控推断新方法[…]
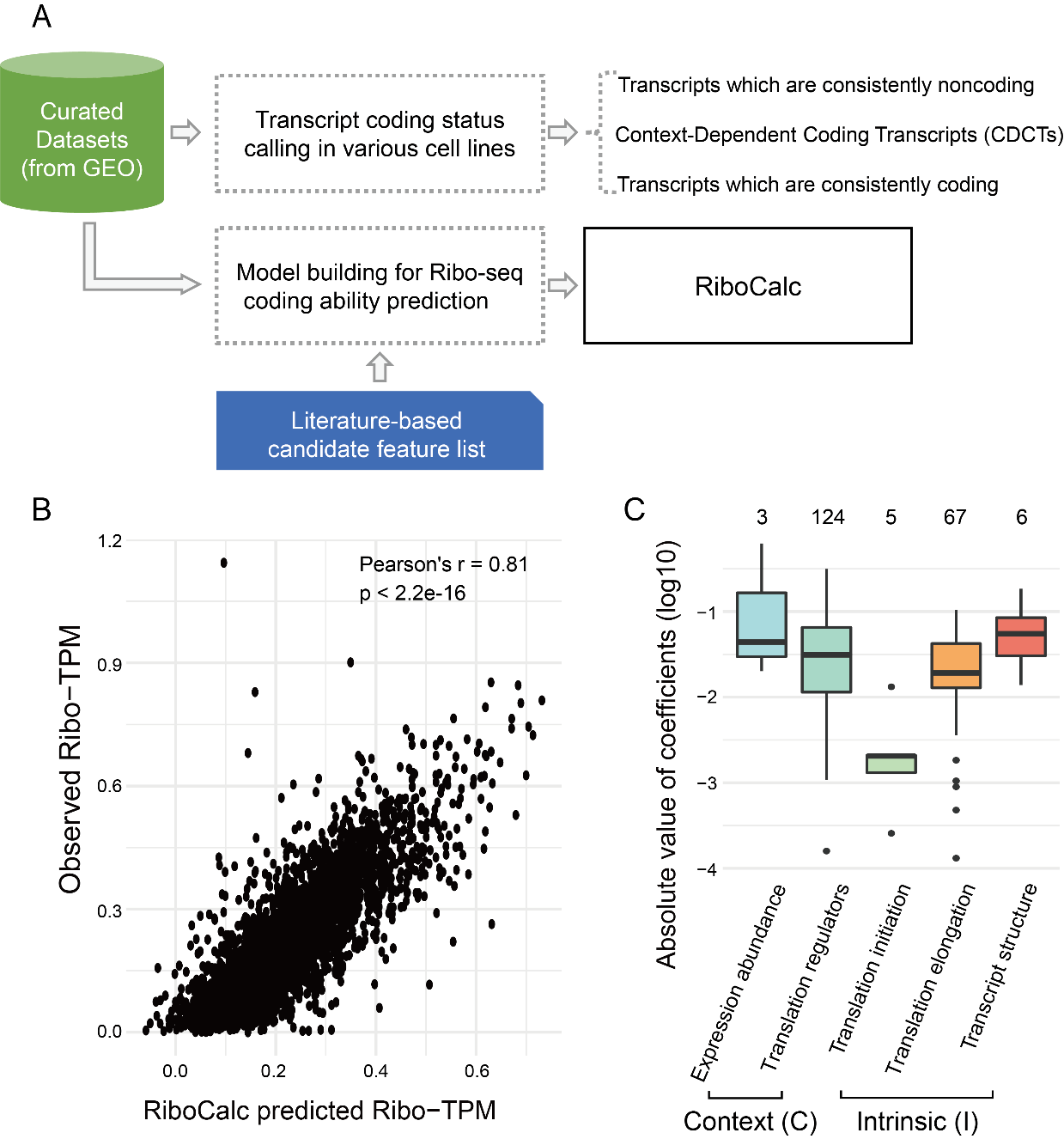
RiboCalc:高歌课题组建立人类RNA转录本编码能力定量预测模型
基因转录在生物学中心法则中起到了承上启下的作用,细胞通过转录-翻译过程多水平调控应对环境压力和疾病。近年来研究 Read more about RiboCalc:高歌课题组建立人类RNA转录本编码能力定量预测模型[…]
热烈欢迎杨仕琦加入本实验室
杨仕琦是本实验室通过昌平实验室招收的第一位职工,目前主要负责单细胞数据平台的搭建。 热烈欢迎杨仕琦的加入! & Read more about 热烈欢迎杨仕琦加入本实验室[…]
热烈欢迎连娴奕同学成为本实验室博后并获得博雅奖学金
热烈欢迎连娴奕同学成为本实验室博士后。 连娴奕同学成功获得了博雅奖学金,可喜可贺!
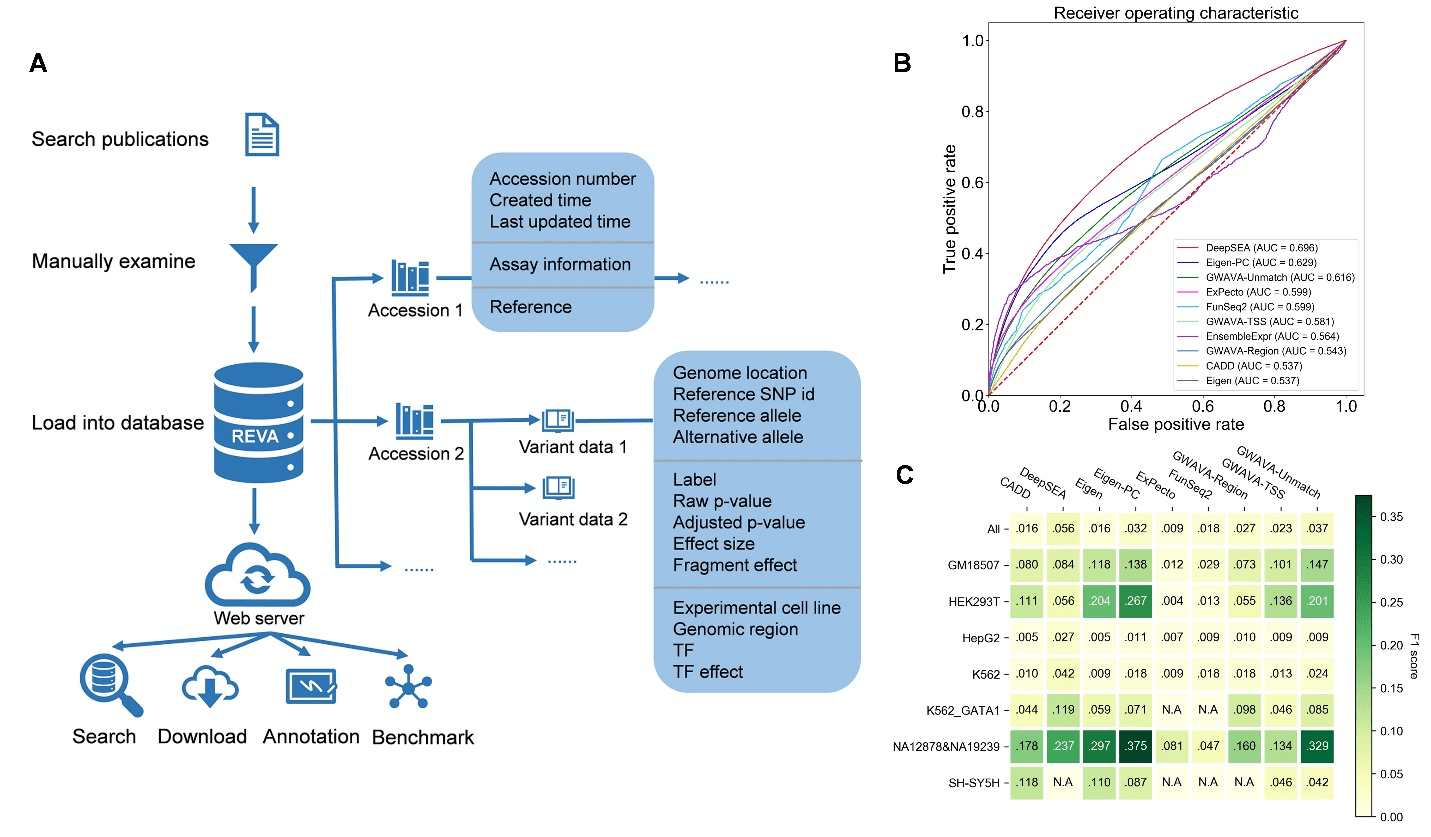
REVA: 高歌课题组在人类调控相关非编码变异整合解析方面取得进展
人类基因组中97%的区域虽不编码蛋白,但仍具有不可忽视的功能,已知超过90%与疾病和性状关联 Read more about REVA: 高歌课题组在人类调控相关非编码变异整合解析方面取得进展[…]
祝贺史方圆同学顺利取得博士学位
史方圆同学在攻读博士学位期间刻苦努力,参与了多项工作,其中代表性的工作为: REVA as A Well-cu Read more about 祝贺史方圆同学顺利取得博士学位[…]