2019年11月8日,北京大学蛋白质与植物基因研究国家重点实验室高歌研究员课题组在Nucleic Acids Research杂志在线发表了题为“PlantRegMap: charting functional regulatory maps in plants”的研究论文(https://doi.org/10.1093/nar/gkz1020)。该研究绘制完成63种植物的功能性转录调控图谱,为深入探究和理解植物转录调控系统如何工作和演变提供了重要的资源。
转录因子通过结合特定的顺式元件调控下游靶基因时空特异地转录,进而精确调控植物发育和快速应对外部胁迫。系统识别植物基因组中的转录因子、调控元件以及二者之间的相互作用对深入理解植物转录系统的工作机理和演变具有至关重要的作用。在之前的工作中,该课题组通过阅览数千篇相关文献整理出一套完整的植物转录因子分类规则,基于此规则从165个植物基因组中系统识别出32万个转录因子,并为每一个转录因子作了详尽的功能和演化注释,构建了植物转录因子数据库PlantTFDB。经过十余年的发展,该植物转录因子分类规则及数据库已被植物领域的研究者广泛使用,相关网站年均外部访问近亿次,多篇论文入选ESI Highly Cited Papers (Top 1%)。
在本项研究中,该团队致力于完成植物转录调控图谱的其余两部分:调控元件及转录因子与调控元件之间的相互作用。首先,他们借助基因组比对构建了63个代表性植物的高精度保守性图谱;然后,基于转录因子结合矩阵各位点的信息熵与对应位点保守性之间的关系开发出一种新算法(FunTFBS)来筛选转录因子与调控元件之间的功能性调控关系;使用上述保守性图谱和算法FunTFBS,系统鉴定出2000余万个功能性转录因子结合位点和200余万功能性转录调控关系,涉及63个物种2万余个转录因子,从而绘制完成覆盖被子植物主要分支的转录调控图谱。至此,该课题组将以转录因子为中心的PlantTFDB升级为一个综合的植物转录调控数据与分析平台PlantRegMap(http://plantregmap.cbi.pku.edu.cn ,镜像 http://plantregmap.gao-lab.org ),为研究和理解植物转录调控系统提供了重要的数据与分析平台。
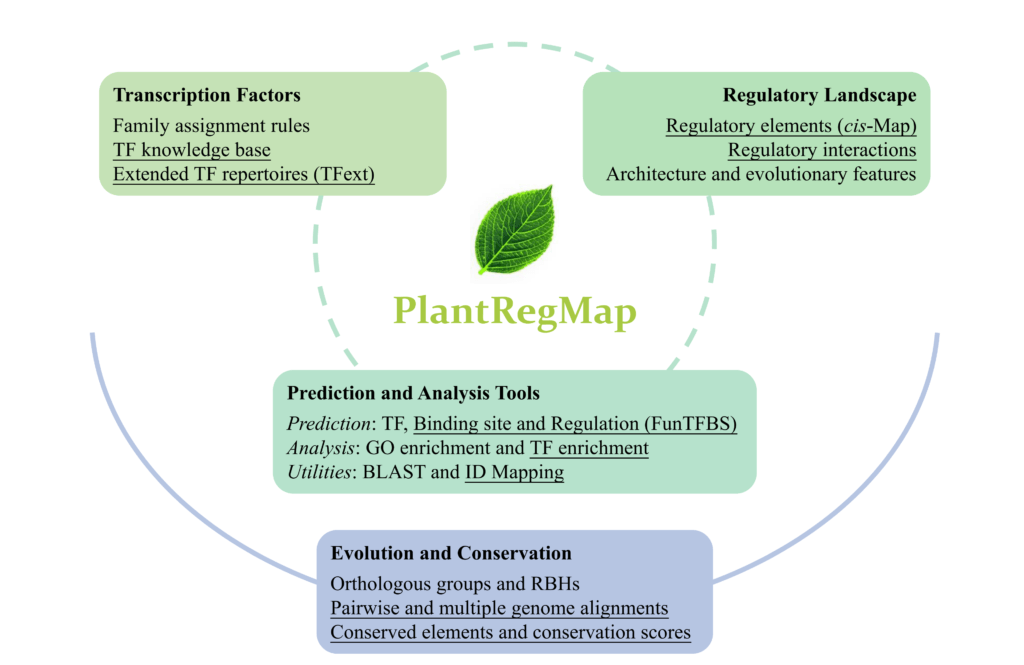
PlantRegMap数据平台的架构(下划线代表在该工作中新发布或更新的模块)
北京大学前沿交叉学科研究院博士研究生田丰、北京大学生命科学学院博士研究生杨德昌为本论文的共同第一作者,北京大学生命科学学院高歌研究员和靳进朴博士为共同通讯作者。该研究得到了国家自然科学基金、北京大学蛋白质与植物基因研究国家重点实验室的经费支持。