高歌研究组2020年发表于《自然·通讯》(Nature Communications)杂志的研究成果《利用Cell BLAST通过无偏细胞嵌入进行大规模scRNA-seq数据库检索》(Searching large-scale scRNA-seq databases via unbiased cell embedding with Cell BLAST),近日经《基因组蛋白质组与生物信息学报》(Genomics, Proteomics and Bioinformatics,简称GPB)评选,入选2020年度“中国生物信息学十大进展”。
作为细胞异质性研究的重要工具,近年来单细胞转录组测序技术蓬勃发展,产生了大量数据。为有效利用这些宝贵数据,高歌团队开发了单细胞转录组数据整合检索方法Cell BLAST。类比于生物序列研究中的BLAST算法,Cell BLAST可以准确快速地对新产生的单细胞数据在已有数据库中进行检索并注释,在节省了传统根据marker基因手动注释所需时间成本的同时,降低了人工操作可能引入的错误。Cell BLAST算法通过对抗学习有效地解决了单细胞转录组检索中复杂的多层次批次效应问题,并基于对单细胞测量过程内在随机性的刻画,提出了一个新的相似性度量指标NPD,可有效应用于单细胞跨数据集整合检索和比较分析。为了充分发挥Cell BLAST的检索能力和作用,团队进一步自主构建了涵盖多组织器官的跨物种单细胞转录组参考数据库ACA,并通过网页提供在线服务(https://cblast.gao-lab.org)。该工作为有效利用现有数据进行细胞注释和跨数据集研究提供了新的工具和资源,也展示了计算生物学、生物信息学方法在复杂生物学体系研究中的强大支撑作用。
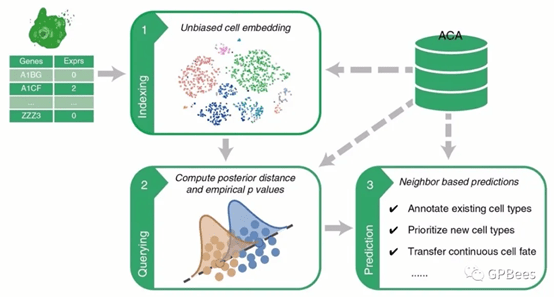
图:单细胞转录组检索方法Cell BLAST的工作流程
Cell BLAST首先将待查询数据与ACA数据库中的参考数据同时映射到低维细胞嵌入空间,在该空间中通过对抗学习消除多层次批次效应,并基于对单细胞测量过程内在随机性的刻画,使用NPD距离查询参考数据中与查询数据最相似的细胞,最后利用查询结果进行多角度自动化数据注释。
中国生物信息学“十大”系列是由《基因组蛋白质组与生物信息学报》于2018年发起组织评选,旨在推动我国生物信息学的学科发展和创新研究,充分展示和宣传我国生物信息学领域的重大研究成果,高歌研究组人类lncRNA研究成果曾入选2019年度“中国生物信息学十大数据库”。
应用和数据库链接:
原文信息:
Cao, Z. J., Wei, L., Lu, S., Yang, D. C. & Gao, G. Searching large-scale scRNA-seq databases via unbiased cell embedding with Cell BLAST. Nat. Commun. 2020; 11:3458. PMID: 32651388